メチル化DNA結合蛋白質MBD4による緩い基質DNA認識
Tweet執筆者情報
執筆者:大谷淳二、有吉眞理子、白川昌宏
連絡先:有吉眞理子 〒615-8501 京都市左京区吉田本町 iCeMS E-mail: aariyoshi@icems.kyoto-u.ac.jp
原著論文:Structural Basis of the Versatile DNA Recognition Ability of the Methyl-CpG Binding Domain of Methyl-CpG Binding Protein 4. The Journal of Biological chemistry 288:6351-6362, 2013.
更新日:2013年4月15日
概要
メチル化CpG結合蛋白質MBD4は、N末端領域にメチル化DNA結合ドメイン(MBD)、C末端領域に塩基除去活性ドメインを持つDNAミスマッチ塩基除去修復酵素である。本研究では、MBD4のMBDドメインの結晶構造に基づき、そのDNA結合表面の柔軟な構造特性が、メチル化DNA、チミン/グアニン(T/G)ミスマッチ部位だけでなく、近年DNA脱メチル化経路の中間体として注目されているメチル化シトシンの酸化誘導体への結合も可能にしていることを明らかにした。今回得られた知見は、ダイナミックなDNAメチル化制御を理解する上で重要な塩基認識機構の新しい一面を明らかにするものである。
***
はじめに
脊椎動物のゲノムのCpG配列中のシトシン塩基のメチル化は、細胞の個性や機能を規定するエピジェネティックな遺伝子発現調節の要である(1)。進化的に保存されたメチル化CpG結合ドメイン(MBD)を持つMBDファミリー蛋白質は、このようなゲノムのメチル化状態を認識し、様々なクロマチン構造変換活性をリクルートすることによって、遺伝子発現抑制因子として機能する。一方、メチル化シトシン塩基は生理条件下での自発的な脱アミノ化などを介して容易にチミン塩基に変換され、突然変異の原因となるT/Gミスマッチを生じる。MBDファミリー蛋白質の一つであるMBD4は、MBDと共に塩基除去活性ドメインを持つT/Gミスマッチ塩基除去修復酵素であり、ゲノム安定性などに重要な役割を果たす(2)。MBD4のMBDは、他のファミリー蛋白質のMBDと類似のアミノ酸配列を保持しているにもかかわらず、メチル化CpGと、脱アミノ化により生じるT/Gミスマッチの両方に結合するというユニークなDNA結合能を有している(2)。本研究では、MBD4の多様なDNA認識機構を明らかにすべく、MBD4のMBD (MBDMBD4)の構造機能解析を行った。
MBD4のMBDはメチル化DNA、ミスマッチDNAと結合する
まず、等温滴定カロリメトリー (ITC) 法を用いて、MBDMBD4のDNA結合特異性の定量的な解析を行った。その結果、MBDMBD4は両鎖メチル化 (5mCG/5mCG)、もしくは、その脱アミノ化産物であるT/Gミスマッチ (5mCG/TG) を含むDNA断片と同等の親和性 (それぞれの解離定数; 97.5 nM, 98.8 nM)で結合することが示された。同じくMBDファミリー蛋白質であるMBD1のMBD (MBDMBD1)の場合、T/Gミスマッチに対する結合親和性は、両鎖メチル化DNAとの親和性の1/10程度であり、二つの塩基配列に対する結合特異性は、MBD4特有の機能であることが確認された。我々は、このようなMBD4にユニークな基質認識を実現する構造要因を理解するため、MBDMBD4と5mCG/TGもしくは5mCG/5mCGを含むDNA断片との複合体の結晶構造を決定した。これら複合体の結晶構造を他のMBDファミリー蛋白質のDNA複合体構造(3,4)と比較すると、骨格構造、及び対称的な5mCG/5mCG配列を認識する二つのアルギニン側鎖 (Arg フィンガー)、を含む基本的なDNA認識モチーフはMBD4においても保存されていた。実際、MBD4-5mCG/5mCG複合体における二つのArg フィンガーの配置は、他のMBDファミリー蛋白質MeCP2の結晶構造とよく一致していた(図1A, C)。
MBD4の柔軟なDNA結合表面が可能にする広い基質特異性
MBD4の二つのArg フィンガーによる5mCG/TGの認識様式は、5mCG/5mCGの場合と大局的には類似している。それでは、なぜMBD4のみが二重の基質特異性を示すのか。MBD4とMeCP2のDNA相互作用表面の構造を詳細に比較すると、水分子を介してメチル化CpGを認識するチロシン残基側鎖の配向の違いと、片方のArg フィンガー (Arg106) の柔軟性という二つの構造要因が基質特異性を説明しうることが判明した(図1)。
一見すると、チミン塩基とメチル化シトシン塩基の化学構造の違いは顕著ではないが、シトシンがメチル化されてもC/G塩基対の水素結合パターンは維持されるのに対して、T/Gミスマッチ塩基対ではWatson-Click型の水素結合が形成できなくなる(図1A, B)。それ故、5mC/G塩基対のメチル化シトシンと比較すると、T/G塩基対中のチミン塩基はDNA二重鎖の主溝側へ1オングストローム程度突出する。MeCP2では、前述のチロシン残基、Tyr123がDNA側を向いているのに対して、MBD4ではTyr96のアミノ酸側鎖がDNA結合表面とは反対側を向いており、DNA結合表面に広い空間が形成されている(図1A, C)。このMBD4に特徴的な空間は、立体障害を引き起こすことなく突出したチミン塩基を受け入れることができる。この空間には蛋白質分子とDNA分子の間を埋めるように水和水が配置されており、メチル化DNAとミスマッチDNAの違いに応じて、水分子間の水素結合のネットワークパターンを変化させている(図1A, B)。
MBDMBD4と典型的なMBDのもう一つの構造的な違いは、片方のArg フィンガー、Arg106の側鎖の柔軟性である。典型的なMBDでは、メチル化DNA認識に重要な二つのArg フィンガーの側鎖は、それぞれの近傍の酸性アミノ酸残基との分子内相互作用によりメチル化DNA認識に有利なコンフォメーションに固定されているが(図1C)、MBD4ではArg106の側鎖を固定する酸性残基を欠いている(図1A, B)。従って、Arg106の側鎖の自由度が獲得され、メチル化DNA、T/Gミスマッチの違いを吸収し、両方の基質と結合できるようになると考えられる。
このようにMBD4のDNA結合表面は、ユニークな構造柔軟性を持っており、それによって結合塩基に対する許容範囲が広くなっていると示唆される。
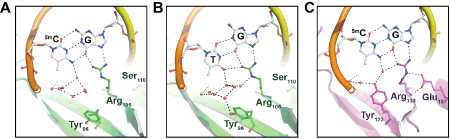
図1. MBD-DNA結合表面の比較:(A)MBD4-5mCG/5mCG複合体、(B)MBD4-5mCG/TG複合体、(C)MeCP2-5mCG/5mCG複合体(参考文献4)水分子を赤い球モデルで、水素結合(>3.2オングストローム)を黒い破線で示している。
MBD4とメチル化シトシンの酸化誘導体の結合
MBDMBD4-DNA結合表面の空間的な自由度から、MBDMBD4は、5位にメチル基よりも大きな官能基が付加されたシトシン塩基も結合することが予測された。近年、高等真核生物におけるDNAの修飾として、5位メチル基がさらに酸化を受けた、ヒドロキシメチルシトシン(hmC)、ホルミルシトシン(foC)、カルボキシシトシン(caC)が細胞内で存在することが明らかになった(5,6, 7)。そこで、これらのメチル化シトシンの酸化誘導体を含むDNAとの結合実験を行うと、MBDMBD1がメチル化DNAにのみ結合する、厳密な特異性を持っていたのと対照的に、MBDMBD4は、hmC、foC、hmUを含むDNAに対しても結合し、寛容な基質認識を示すことが明らかになった。そこで、MBDMBD4とhmC塩基を含むDNA(5mCG/hmCG)の複合体結晶構造解析を行うと、DNA結合表面の水和水のネットワークが変化して、hmC塩基のヒドロキシ基と水素結合を形成している様子が明らかになった(図2)。
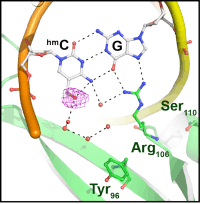
図2. MBD4-5mCG/hmCG複合体のDNA結合表面: 水分子を赤い球モデル、水素結合(>3.2オングストローム)を黒い破線、hmC塩基のヒドロキシ基の電子密度を赤いメッシュで示している。
まとめ
MBDMBD4とDNA断片の結晶構造解析から、MBDMBD4の寛容な塩基認識を支える2つの特徴、(i)DNA結合表面の空間に可塑的な水和水のネットワーク形成、(ii)Arg106の側鎖の柔軟性、が明らかになった。MBDMBD4の寛容なDNA認識特異性は、MBD4が転写抑制とT/Gミスマッチ修復という複数の機能を果たすことを反映していると考えられる。また、MBD4の結合基質には、hmC、foC、hmUを含むDNA断片も含まれることが明らかになったが、MBD4の塩基除去実験では、T/G、U/G、hmU/Gといった、ミスマッチ塩基対のみが基質として切除された。hmC、foC、caC塩基は、現在DNA脱メチル化の中間体として非常に注目されているが、細胞内でこれらの塩基は長寿命であるという観測もあり(8,9)、MBD4が酸化シトシンのシグナルを読み取るリーダーとして働いている可能性もある。酸化シトシン塩基の機能におけるMBD4の役割については今後の研究が待たれる。
***
参考文献
1. Bird A (2002) Genes Dev 16: 6-21
2. Hendrich B et al. (1999) Nature 401:301-304
3. Ohki I et al. (2001) Cell 105: 487-497
4. Ho KL et al. (2008) Mol Cell 29: 525-531
5. Kriaucionis S et al. (2009) Science 324:929-930
6. Tahiliani M et al. (2009) Science 324:930-935
7. He YF et al. (2011) Science 333:1303-1307
8. Inoue A et al. (2011) Cell Res 21:1670-1676
9. Inoue A et al. (2011) Science 334:194

